日新月異的生物序列次世代定序技術、單細胞基因分析、經濟作物的全基因體解析與個人化精準醫學等,將生物醫學研究推進至巨量資料層級,特別是個人的基因體組裝(正常與癌化組織),伴隨巨量生物序列的累積,將能揭露更多致病機制與提供未來治療的可能策略,讓我們人類有機會邁向更為健康長壽的新境界。
2019年十月三十日由行政院所積極推動的「台灣精準醫療Biobank整合平台」,將帶領台灣個人化精準醫學,與個人基因體定序分析相關研究的大步邁進。然而,對於混合不同平台、不同品質與定序長度所產出之大量序列,來進行高品質的基因體重組(de novo genome assembly),目前並沒有很好的工具程式。由於基因定序平台的快速演進與多樣化,定序長度已由原來的數百鹼基,進展到五千以上甚至數十萬的鹼基長度,同時一些與染色體結構的相關定序技術(如Hi-C等),也提供染色體層級的對應資訊。此外新的策略如Bionano Genome Mapping Systems等,以光學方式重新進行染色體基因體長片段測繪(Genome Mapping)的方式,可以讓我們得以瞭解染色體層級的真實排列順序與大片段的基因結構差異。然而,現今既存之基因重組演算法大多無法結合不同定序策略的優點,少數可用之方法的組裝結果不佳且速度緩慢。針對這樣的現況,必需發展新一代的快速高效率演算法,利用雲端平台與分散式運算的優勢,才能將結合二代定序、三代定序及基因體光學解析等技術,將基因體組裝推進至染色體層級。以更為完整的面貌,來解析複雜的基因上下游調控機制、找尋個人精準治療策略與協助基因體育種等之複雜課題。然而,如何在有限的定序經費與計算資源下,在定序策略與組裝品質之間取得平衡,是基因體研究領域當今所遇到的最大挑戰。
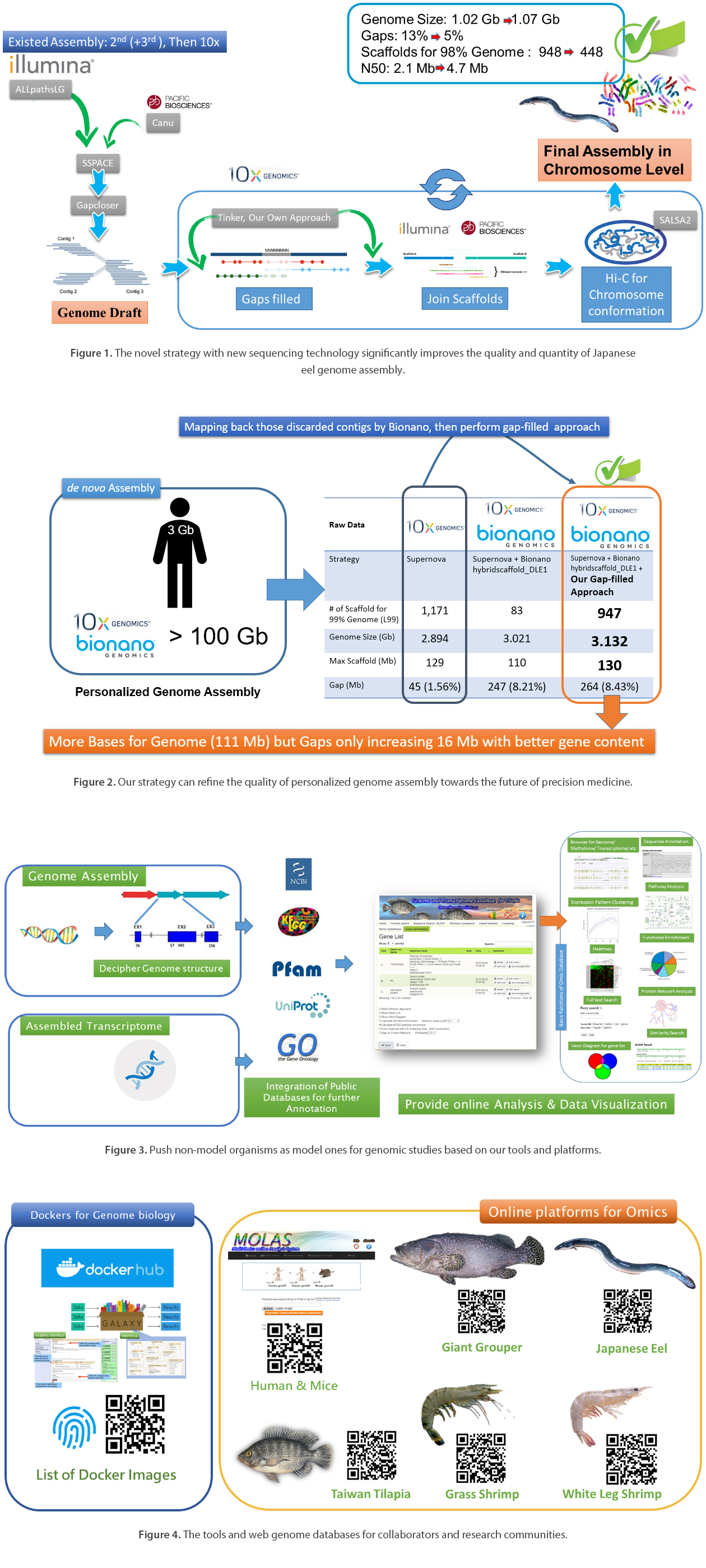
目前研究團隊透過手邊已有大量數TB的高品質的自產資料(包括次世代定序資料、第三代單分子定序及10x Genomics短片段標誌序列等),研發了新的混合組裝演算法。除了能在組裝前後進行序列的分析,也能動態地以遞迴(Spiral assembling)與多樣定序平台混搭的方式,來優化整個組裝的基因體,同時也能利用大量的10x Genomics短片段標誌序列,來重新組裝與修補在原有序列中的遺缺,以及透過組裝序列間的拓樸關係,來連結與修補可能相鄰的長序列大片段。目前在日本鰻、台灣鯛及個人基因體組裝上,已有相當不錯的進展,能大幅減少序列中的遺缺,並連結組裝片段,同時增加基因內涵。
在非模式物種方面,對於已有不同平台定序資料的之雛形基因體,我們已發展可動態組合的演算法,依其定序量與類型,來找出最佳的組裝方式。對於全新的定序計畫,我們則建議直接以三代定序為主,結合加上標誌序列的二代定序 (以10x Genomics所產出的短序列標誌連鎖組)與染色體構象Hi-C定序為輔,在考量定序成本與基因體覆蓋倍率的平衡下,利用所開發新一代組裝演算法模組,將可提供全新高品質基因體組裝新策略。基於前項的成功經驗,已有院內生多中心、台大、海大與台灣水產試驗所等團隊與我們接觸商討合作,並簽訂MOU,希望透過我們的經驗,來完成複雜的基因體解碼工作(如白蟻、海水台灣鯛、白帶魚及金目鱸等),作為後續基因體育種的重要基礎(圖一)。
在個人化基因體重新組裝部分,將與中研院生物醫學研究所團隊合作,整合10x Genomics與bionano光學測繪的大量資料(一個人約有超過100Gb 以上的數據),來發展新一代雲端組裝演算法,建構高品質個人基因體。目前已能有效地將原有組裝之遺缺(Gaps)部分大幅縮減,並增加序列長度與基因內容,也就是提昇整體組裝的解析度與質量。將能作為未來結合人工智慧與相關醫療資料後,發展精準醫療的重要紮實基礎(圖二)。同時,為了解決大量運算所造成的計算需求,在高效率雲端計算流程建構部分,研發團隊將與台灣微軟公司一同合作開發。
本研究之相關成果也陸續整理釋出程式源碼及建構Dockers images,放置在GitHub及 Docker Hub 上。組裝出來的經濟生物基因體,也透過研發團隊所建構的平台,進行註解與線上資料庫建構,將這一些非模式物種之基因體研究,提昇到如同果蠅或小鼠等模式生物的水準,協助相關研究社群進行更詳盡的研究(圖三、四)。同時,我們也透過參與國際研討會與舉辦工作坊的方式,以及一年一度的院區開放日來宣傳與推廣。讓更多院內外的生物醫學研究社群得以善用這些工具,更深入他們自身的研究主題。此外,研發成果中的『人類與小鼠基因表現分析平台』,已與國內定序廠商合作。以技術授權的方式完成轉移威健生物科技股份有限公司,於2018年底開始商業試運轉。
